Richard Preiss1,2, Caroline Tyrawa1, Kristoffer Krogerus3,4, Lars Marius Garshol5 and George van der Merwe1*La producción generalizada de alimentos y bebidas fermentadas ha resultado en la domesticación de levaduras de Saccharomyces cerevisiae específicamente adaptadas a la producción de cerveza. Si bien hay evidencia de que la industrialización de la cerveza aceleró la domesticación de la levadura de cerveza, también existe un cultivo cervecero artesanal en el oeste de Noruega que ha transmitido levaduras denominadas kveik durante generaciones. Esta práctica ha resultado en levaduras ale que son típicamente altamente floculantes, fenólicas de sabor negativo (POF-), y exhiben una alta tasa de fermentación, similar a los linajes previamente caracterizados de levadura domesticada. Además, las levaduras kveik son, según informes, tolerantes a altas temperaturas, probablemente debido a la práctica tradicional de verter la levadura en mosto caliente (> 28 ° C). Aquí, caracterizamos las levaduras kveik de 9 fuentes noruegas diferentes mediante la toma de huellas dactilares por PCR, la secuenciación del genoma completo de cepas seleccionadas, las pantallas fenotípicas y las fermentaciones a escala de laboratorio. El análisis filogenético sugiere que las levaduras kveik forman un grupo distinto entre las levaduras de cerveza. Además, identificamos una nueva mutación POF-pérdida de función, así como SNP y CNV potencialmente relevantes para la termotolerancia, tolerancia alta al etanol y fenotipos de alta velocidad de fermentación de cepas kveik. También identificamos los marcadores de domesticación relacionados con la floculación en kveik. Tomados en conjunto, los resultados sugieren que las levaduras kveik noruegas son un grupo genéticamente distinto de levaduras de cerveza domesticadas con propiedades altamente relevantes para el sector cervecero.
1Department of Molecular and Cellular Biology, University of Guelph, Guelph, ON, Canada
2Escarpment Laboratories, Guelph, ON, Canada
3VTT Technical Research Centre of Finland, Espoo, Finland
4Department of Biotechnology and Chemical Technology, School of Chemical Technology, Aalto University, Espoo, Finland
5Independent Researcher, Rælingen, Norway
Introducción
Está claro que la actividad humana resultó en la domesticación de levaduras de Saccharomyces cerevisiae específicamente adaptadas para la producción de cerveza. Recientemente, se ha demostrado que las levaduras de cerveza industriales actuales se originaron a partir de un puñado de ancestros domesticados, con un clado mayor, "Cerveza 1", que comprende la mayoría de las levaduras ale alemanas, británicas y estadounidenses, y otro clado ". Beer 2, "que no tiene una estructura geográfica y está más relacionada con las levaduras de vino" (Gallone et al., 2016). En general, parece que la selección humana de levaduras de cerveza en el transcurso de los siglos ha dado lugar a la evolución de mecanismos para: fermentar eficazmente azúcares de mosto tales como maltosa y maltotriosa a través de duplicaciones de genes MAL; eliminar la producción de fenol off flavor (POF) por mutaciones sin sentido frecuentes en los genes PAD1 y FDC1, responsables de la producción de 4-vinylguaiacol (4-VG), generando así cepas POF negativas (POF-), y; floculan de manera eficiente, ayudando así en el procesamiento posterior del producto (McMurrough et al., 1996; Brown et al., 2010; Steensels y Verstrepen, 2014; Gallone et al., 2016; Gonçalves et al., 2016).
Independientemente de la región de origen, la levadura de cerveza probablemente se mantuvo y domesticó por reutilización (repitching) y se compartió entre generaciones de cerveceros, lo que resultó en muchas de las levaduras de cerveza domesticadas utilizadas en la actualidad (Gibson et al., 2007; Libkind et al., 2011; Steensels et al., 2014; Gallone et al., 2016). No debe suponerse, sin embargo, que la domesticación de las levaduras de cerveza ocurrió únicamente dentro de los límites de las cervecerías industriales, ya que existían tradiciones cerveceras anteriores a la industrialización de la cerveza en el norte de Europa (Nordland, 1969; Räsänen, 1975). Estos cerveceros usaban cepas de levadura que mantenían por sí mismas, y la misma levadura generalmente se usaba para preparar y hornear. Sin embargo, en Noruega y Suecia, la cerveza y los panes sin levadura son anteriores al pan con levadura debido a la falta de grano adecuado (Visted y Stigum, 1971). Las mejoras en el transporte y la creciente especialización económica causaron que la elaboración tradicional de la granja disminuyera a partir del siglo XIX, lo que junto con la entrada de la levadura comercial probablemente condujo a la desaparición de muchas levaduras cerveceras tradicionales (Nordland, 1969).
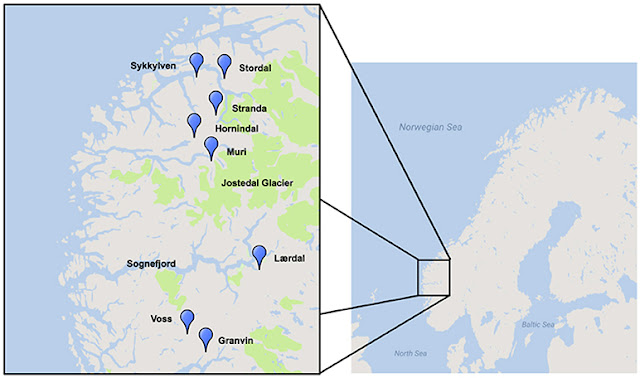
Una región donde todavía se utilizan cultivos de levadura tradicionales es el oeste de Noruega, donde una serie de cerveceros de granja han mantenido las levaduras tradicionales de esta región, algunas según se informa durante cientos de años (Figura 1; Nordland, 1969). La cerveza de granja noruega se produce predominantemente a partir de cebada malteada y normalmente se salta, y también se infunde con ramas de enebro (Nordland, 1969). Las cervezas de las granjas se conocen como maltøl o kornøl. Hasta hace poco, los cultivos de levadura, conocidos como kveik, un término dialectal para la levadura en esta región, estaban geográficamente aislados y mantenidos localmente solo por los cerveceros tradicionales. Se hipotetiza que las levaduras kveik son domesticadas, ya que las cervezas producidas usando estas levaduras son no fenólicas (POF-) y estas levaduras son potencialmente capaces de fermentar rápidamente los azúcares derivados de la malta debido a los breves tiempos de fermentación informados. Además, al igual que las levaduras de cerveza domesticadas, las levaduras kveik se mantienen y se vuelven a utilizar mediante el repicado serial (Gibson et al., 2007; Garshol, 2014; Stewart, 2015).
Sorprendentemente, los registros de levadura, creados específicamente para el almacenamiento de kveik, pueden fecharse al menos desde AD 1621 (Nordland, 1969), lo que sugiere que la reutilización de kveik comenzó mucho antes de esta fecha, ya que presumiblemente la levadura se reutilizaba antes de la desarrollo de tecnología especializada para almacenamiento de levadura. Esto se alinea con, y potencialmente es anterior a, el modelado predictivo reciente de la línea de tiempo de la domesticación de levadura moderna alrededor de A.D. 1573-1604 (Gallone et al., 2016). Por lo tanto, Kveik puede ser un grupo de levaduras de cerveza que han sido domesticadas y mantenidas por una tradición cervecera aislada geográficamente, paralela a la producción de cerveza industrial.
Sin embargo, críticamente poco se entiende sobre las levaduras kveik. Si bien algunas de estas levaduras ahora se han compartido a nivel mundial, hay una falta de datos fenotípicos y genotípicos empíricos pertenecientes a este intrigante grupo de levaduras de cerveza. Aquí informamos la toma de huellas dactilares de PCR y los datos de la secuencia del genoma completo que sugieren que las levaduras kveik forman un grupo interrelacionado de levaduras de cerveza genéticamente distintas de las levaduras de cerveza domesticadas conocidas. Nuestras caracterizaciones fenotípicas y la secuenciación del genoma completo revelan evidencia de domesticación y características positivas en la producción de compuestos saborizantes y la tolerancia al estrés que sugiere el potencial de las levaduras kveik en una amplia gama de aplicaciones industriales.
Materiales y métodos
Cepas de levadura
En el estudio se analizaron un total de 9 muestras de kveik noruego y una muestra adicional de levadura de granja lituana. Siete kveik se suministraron como suspensiones líquidas, y dos se suministraron como muestras de levadura secas. Las muestras secas se rehidrataron en agua estéril. Las suspensiones líquidas de levadura se enriquecieron inoculando 50 μl de la suspensión en 5 ml de YPD (extracto de levadura al 1%, peptona al 2%, dextrosa al 2%). Las muestras se incubaron a 30 ° C durante 24 h con agitación, luego se colocaron en placas en agar Wallerstein Nutrient (WLN; Thermo Fisher CM0309), un medio diferencial para levaduras que distingue a las múltiples levaduras entre sí en una muestra sobre la base de la absorción de el tinte verde de bromocresol. Las colonias de levadura se substirieron a WLN para garantizar la pureza. Las cepas resultantes se resumen en la Tabla 1. Cepas de control adicionales para los experimentos se enumeran en la Tabla 1.
 |
| Tabla 1. Cepas de levadura investigadas, información de origen e identificación de secuencia. |
El ADN se extrajo utilizando una adaptación de un método descrito anteriormente (Ausubel et al., 2002). Brevemente, se cultivaron células de levadura en 3 ml de caldo YPD a 30 ° C, 170 rpm durante 24 h, se lavaron con agua estéril y se sedimentaron. Las células se resuspendieron en 200 μl de tampón de ruptura (2% de Triton X-100, 1% de SDS, 100 mM de NaCl, 10 mM de Tris-HCl). Se añadieron 0,3 g de perlas de vidrio y 200 \ mu l de fenol / cloroformo / alcohol isoamílico y las muestras se sometieron a agitación vorticial continuamente a velocidad máxima durante 3 min para lisar las células. Después de la centrifugación, la capa acuosa se transfirió a un tubo limpio y se añadió 1 ml de etanol al 100%. El sobrenadante se eliminó después de otra etapa de centrifugación. El sedimento resultante se resuspendió en 400 μL de tampón TE 1X y 30 μL de RNasa A libre de DNasa 1 mg / mL y se incubó a 37 ° C durante 5 min. El sedimento se lavó entonces con 1 ml de etanol al 100% y 10 μl de acetato de amonio 4 M, seguido de otro lavado con 1 ml de etanol al 70%, y luego se resuspendió en 100 μl de ddH2O estéril.
Secuenciación de PCR e ITS
Las regiones espaciadoras transcritas internamente (ITS) de las cepas de levadura se amplificaron usando cebadores ITS1 e ITS4 (Pham et al., 2011). Las reacciones de PCR contenían 1 \ mu l de ADN genómico, 2,5 \ mu M de cada cebador, dNTP 0,4 mM, 2,5 U de ADN polimerasa Taq y tampón de reacción Taq 1X. Las reacciones de amplificación se llevaron a cabo en un Termociclador BioRad T100 bajo las condiciones descritas previamente (Pham et al., 2011). Los productos de PCR se visualizaron en un gel de agarosa al 1% en tampón TAE 1X para confirmar la amplificación con éxito. Las muestras se purificaron usando el kit de purificación QIAquick PCR y se secuenciaron usando un analizador de ADN Applied Biosystems 3730. El software 4peaks se usó para realizar un control de calidad de los rastros de secuencia. Las secuencias resultantes se analizaron para homología a nivel de especie usando NCBI BLAST (blastn suite).
Huella de ADN
Las cepas de levadura se identificaron por interdelta PCR fingerprinting utilizando cebadores interdelta δ2 (5'-GTGGATTTTTATTCCAACA-3 '), δ12 (5'-TCAACAATGGAATCCCAAC-3') y δ21 (5'-CATCTTAACACCGTATATGA-3 ') (Ness et al., 1993; Legras y Karst, 2003). Los pares de cebadores seleccionados para una mayor amplificación y análisis fueron δ2 + δ12 y δ12 + δ21, que produjeron el mayor rango de bandas bien resueltas. La PCR se llevó a cabo de la siguiente manera: 4 min a 95 ° C, luego 35 ciclos de 30 s a 95 ° C, 30 s a 46 ° C, luego 90 s a 72 ° C, seguido de un paso final de 10 min a 72 ° C (Legras y Karst, 2003). Los productos de reacción se confirmaron mediante electroforesis en un gel de agarosa al 1% en tampón TAE 1X. Las muestras de PCR se purificaron luego usando un kit de purificación QIAquick PCR y se analizaron en un Bioanalizador Agilent 2100 usando el chip Agilent DNA 7500. Los patrones de bandas obtenidos usando Bioanalyzer se analizaron usando el software GelJ (Heras et al., 2015). Las comparaciones para cada conjunto de cebadores (δ2 + δ12 y δ12 + δ21) se generaron de forma independiente utilizando la característica de comparación del software, agrupando las huellas dactilares utilizando la correlación de Pearson y UPGMA (Heras et al., 2015). Las matrices de distancia individuales resultantes se combinaron usando fuse.plot en R (https://github.com/andrewfletch/fuse.plot), que usa el algoritmo hclust para formatear y fusionar las matrices y realizar clusters jerárquicos con UPGMA. Los datos se visualizaron utilizando el software FigTree (http://tree.bio.ed.ac.uk/software/figtree/).
Contenido de ADN por citometría de flujo
La citometría de flujo se realizó en seis cepas kveik para estimar la ploidía esencialmente como se describe por Haase y Reed (2002). Las células se cultivaron durante la noche en medio YPD, y 1 x 107 células ~ se lavaron con 1 ml de 50 mM de tampón de citrato. Las células se fijaron luego con etanol al 70% frío, y se incubaron durante la noche a -20 ° C. Después, las células se lavaron con tampón de citrato 50 mM (pH 7,2), se resuspendieron en tampón de citrato 50 mM que contiene 0,25 mg ml-1 de ARNasa A y se incubaron durante la noche a 37 ° C. Después, se añadió 1 mg ml-1 de proteinasa K, y las células se incubaron durante 1 hora a 50 ° C. Después, las células se tiñeron con SYTOX Green (2 μM, Life Technologies, EE. UU.), Y su contenido de ADN se determinó usando un citómetro FACSAria IIu (Becton-Dickinson, EE. UU.). Los contenidos de ADN se estimaron comparando las intensidades de fluorescencia con las cepas de referencia haploides de S. cerevisiae (CEN.PK113-1A) y diploides (CEN.PK). Se recolectaron cien mil eventos por muestra durante la citometría de flujo. Los datos se procesaron con el paquete "flowCore" (Hahne et al., 2009) en R, mientras que las intensidades medias de fluorescencia máxima se estimaron con la función "normalmixEM" del paquete "mixtools" (Benaglia et al., 2009) en R.
Secuenciación y Análisis del Genoma
Los genomas completos de ocho cepas (seis cepas kveik y dos cepas cerveceras comerciales como controles, véase la Tabla 1) fueron secuenciados por Genome Québec (Montreal, Canadá). En resumen, se aisló el ADN como se describió anteriormente, después de lo cual se preparó una biblioteca Illumina TruSeq LT de 150 pares de bases por pares para cada cepa y la secuenciación se llevó a cabo con un instrumento HiSeqX. Las lecturas de secuenciamiento se analizaron con calidad con FastQC (versión 0.11.5) (Andrews, 2010) y se recortaron y se filtraron con Trimmomatic (versión 0.36; consulte la Tabla complementaria S2 para conocer los parámetros) (Bolger et al., 2014). Las lecturas fueron alineados a un genoma de S. cerevisiae S288c (R64-2-1) de referencia utilizando SpeedSeq (0.1.0) (Chiang et al., 2015). La calidad de las alineaciones se evaluó con QualiMap (2.2.1) (García-Alcalde et al., 2012). El análisis de variantes se realizó en lecturas alineadas utilizando FreeBayes (1.1.0-46-g8d2b3a0l; consulte la Tabla Suplementaria S2 para conocer los parámetros) (Garrison y Marth, 2012). Las variantes en todas las cepas se llamaron simultáneamente (multi-muestra). Antes del análisis de variante, las alineaciones se filtraron a un MAPQ mínimo de 50 con SAMtools (1.2; consulte la Tabla S2 complementaria para ver los parámetros) (Li et al., 2009). Anotación y efecto de predicción de las variantes se realizó con SnpEff (1.2; ver el cuadro complementario S2 para los parámetros) (Cingolani et al, 2012).. Se estimaron las variaciones en el número de copias de los cromosomas y los genes en función de la cobertura con Control-FREEC (11.0; consulte la Tabla Suplementaria S2 para conocer los parámetros) (Boeva et al., 2012). Se identificaron variaciones en el número de copias estadísticamente significativas usando la prueba de suma de rangos de Wilcoxon (p <0.05). La cobertura mediana y el recuento de SNP heterocigotos en ventanas de 10.000 pb se calcularon con BEDTools (2.26.0) (Quinlan y Hall, 2010) y se visualizaron en R.
Análisis filogenético y de la estructura de la población
Antes del análisis filogenético y de la estructura de la población, se llamaron genotipos de consenso para las cepas secuenciadas a partir de las variantes identificadas utilizando BCFtools (1.2) (Li, 2011). Debido a los altos niveles de heterocigosidad (> 50,000 SNP heterocigóticos) en las seis cepas kveik, también se intentó la eliminación del haplotipo utilizando WhatsHap (0.14.1) (Martin et al., 2016). WhatsHap es una herramienta de fase basada en lectura, que utiliza lecturas de secuencia mapeadas que abarcan al menos dos variantes heterocigóticas para inferir la fase. Los haplotipos de consenso se invocaron a partir de las variantes de fase utilizando BCFtools. Conjuntos de genoma de las 157 cepas de S. cerevisiae descritas en Gallone et al. (2016) fueron recuperados de NCBI (BioProject PRJNA323691). Además, el ensamblaje del genoma de Saccharomyces paradoxus CBS432 se obtuvo de https://yjx1217.github.io/Yeast_PacBio_2016/data/ (Yue et al., 2017) para ser utilizado como un grupo externo. Múltiples alineamientos de secuencia de los genotipos de consenso de las ocho cepas secuenciadas y los 158 conjuntos se realizó con la tubería NASP (1.0.0) (Roe et al., 2016) utilizando S. cerevisiae S288c (R64-2-1) como referencia genoma Se extrajo una matriz de polimorfismos de un solo nucleótido (SNP) en las 167 cepas de las secuencias alineadas. Los SNP se anotaron con SnpEff (Cingolani et al., 2012) y se filtraron de la siguiente manera: solo se conservaron los sitios que estaban en la secuencia codificante de genes, presentes en las 167 cepas y con una frecuencia de alelos minoritarios> 1% (una cepa) . La matriz filtrada contenía 4161584 SNP (142120 sitios). Se estimó un árbol filogenético de máxima verosimilitud utilizando IQ-TREE (1.5.5, ver Tabla Suplementaria S2 para los parámetros) (Nguyen et al., 2015). IQ-TREE se ejecutó utilizando el modelo "GTR + F + R4" y 1000 réplicas de arranque ultrarrápido (Minh et al., 2013). El árbol de máxima verosimilitud resultante se visualizó en iTOL (Letunic y Bork, 2016) y se enraizó en S. paradoxus CBS432. Los pasos anteriores de alineación de secuencia múltiple en adelante se repitieron con los haplotipos de consenso en fase de las seis cepas kveik.
La estructura de la población de 165 cepas se investigó utilizando los algoritmos basados en modelos en ESTRUCTURA (2.3.4, ver Tabla Suplementaria S2 para los parámetros) (Pritchard et al., 2000) y fastStructure (1.0; ver Tabla Suplementaria S2 para los parámetros) (Raj et al., 2014). Ambas herramientas se ejecutaron en múltiples hilos usando structure_threader (1.2.4; ver la Tabla Suplementaria 2 para los parámetros) (Pina-Martins et al., 2017). La matriz de SNP producida a partir del alineamiento de secuencia múltiple se filtró utilizando PLINK (1.9; consulte la Tabla Suplementaria S2 para conocer los parámetros) (Purcell et al., 2007) mediante la eliminación de sitios en desequilibrio de ligamiento (usando un tamaño de ventana de 50 SNP, 5 pasos de SNP). y umbral por pares de 0.5) y con una frecuencia de alelos menores <5%. Además, los SNP de S. cerevisiae S288c y S. paradoxus CBS432 se excluyeron del análisis de la estructura de la población. La matriz SNP adelgazada, que ahora consta de 26583 sitios, se usó como entrada tanto para STRUCTURE como para fastStructure, que se ejecutaron para 1 a 11 poblaciones ancestrales (K). La matriz SNP está disponible como la Hoja de datos suplementarios 1. El algoritmo ESTRUCTURA se ejecutó en 10 réplicas independientes para cada valor K y con un período inicial de grabación de 100.000 iteraciones, seguido de 100.000 iteraciones de muestreo. El número de poblaciones ancestrales (K) que mejor representaba este conjunto de datos se eligió según el "método Evanno" (Evanno et al., 2005; Earl y vonHoldt, 2012) para los resultados de ESTRUCTURA con ESTRUCTURA COSECHADORA y por el valor K que maximizó probabilidad marginal para los resultados de fastStructure (Raj et al., 2014). Los resultados de ESTRUCTURA finalmente se agruparon con el servidor CLUMPAK en línea (Kopelman et al., 2015). Los resultados se trazaron en parcelas tipo "distruct" en R. El análisis del componente principal de la matriz SNP adelgazada producida para el análisis de estructura de la población también se realizó usando el paquete SNPRelate (Zheng et al., 2012). Las diversidades de nucleótidos dentro y entre las poblaciones se estimaron en R utilizando el paquete PopGenome (Pfeifer et al., 2014).
Preparación del mosto
El mosto utilizado para la fermentación de la cerveza y la propagación de la levadura se obtuvo de una cervecería comercial, Royal City Brewing (Guelph, ON). El mosto con lúpulo fue preparado usando malta canadiense de 2 hileras con una gravedad original de 12.5 ° de Platón (1.050 de gravedad específica). El mosto se esterilizó antes de su uso a 121 ° C durante 20 min, y se enfrió a la temperatura de propagación o fermentación deseada durante la noche.
Propagación y fermentación
Las colonias de placas WLN se inocularon en 5 ml de YPD y se cultivaron a 30 ° C, 170 rpm durante 24 h. Los cultivos de YPD se transfirieron a 50 ml de mosto esterilizado y se cultivaron a 30 ° C, 170 rpm durante 24 h. Estos cultivos se contaron usando un hemocitómetro y se inocularon a una velocidad de 1,2 × 107 células / ml en 50 ml de mosto esterilizado en "frascos de especias" de vidrio (frascos de vidrio con un volumen total de 100 ml con lados rectos) provistos de cámaras de aire. Estas fermentaciones a pequeña escala se realizaron por triplicado a 30 ° C durante 12 días. Se eligió una temperatura de 30 ° C como la temperatura de fermentación, ya que es una temperatura común en la fabricación noruega de la granja (Tabla S1). Los tarros se incubaron sin agitar para aproximarse mejor a las condiciones típicas de fermentación de la cerveza. Los perfiles de fermentación se obtuvieron pesando los frascos de especias para medir la pérdida de peso, normalizándose frente a la evaporación del agua de las cámaras de aire.
Análisis de metabolitos de cerveza
Después de la fermentación, las muestras se recogieron y filtraron con filtros de jeringa de 0,45 μm antes del análisis de metabolitos. El análisis del metabolito del sabor se realizó utilizando HS-SPME-GC-MS (Rodríguez-Bencomo et al., 2012). Las muestras contenían 2 mL de cerveza, 0.6 g de NaCl, 10 μL de 3-octanol (0.01 mg / mL) y 10 μL de 3,4-dimetilfenol (0.4 mg / mL). 3-octanol y 3,4-dimetilfenol se usaron como estándares internos. El contenido de etanol y azúcar se midió usando HPLC y un detector de índice de refracción (RI). Las muestras se analizaron usando una columna Aminex HPX-87H, usando ácido sulfúrico 5 mM como fase móvil, en las siguientes condiciones: caudal de 0,6 ml / min, 620 psi y 60 ° C. Cada muestra contenía 400 μL de cerveza filtrada y 50 μL de isopropanol al 6% (v / v) como patrón interno.
Ensayos fenotípicos
Para determinar la tolerancia a la temperatura, la levadura cultivada durante 24 ha 170 rpm a 30 ° C en YPD se subcultivó en YPD precalentada a temperaturas específicas (30, 40, 42, 43, 45 ° C) por duplicado a una DO600 inicial de 0.1 e incubar con agitación durante 20 ha la temperatura indicada. Para determinar la tolerancia al etanol, los cultivos de levadura cultivados durante 24 ha 170 rpm a 30 ° C en YPD se subcultivaron en YPD que contenía concentraciones crecientes de etanol (YPD + EtOH 10, 12, 14, 15, 16%) por duplicado en un OD600 inicial de 0,1 e incubado con agitación durante 20 ha la temperatura indicada. Para evaluar el rendimiento de crecimiento para la tolerancia a la temperatura y el etanol, las muestras de levadura se sometieron a un decolorado usando ácido fosfórico y se tomaron medidas inmediatas de OD600 usando un espectrofotómetro (Simpson y Hammond, 1989). Para determinar la floculación, se cultivaron cultivos de levadura durante 24 ha 170 rpm a 30 ° C en YPD, y luego se inocularon 0,5 ml en 5 ml de mosto esterilizado, que se incubó durante 24 ha 170 rpm a 30 ° C. La floculación se evaluó utilizando la metodología de absorbancia espectrofotométrica del método ASBC Yeast-11 (ASBC, 2011). Los valores se expresan como% de floculación, con <20% representando levadura no floculante y> 85% representando levadura altamente floculante.
Análisis estadístico
El análisis estadístico se realizó en la fermentación, metabolito y datos fenotípicos con ANOVA de una vía y la prueba de Tukey usando el paquete "agricolae" en R (http://www.r-project.org/). Los resultados de las pruebas estadísticas están disponibles como Hoja de datos complementarios 2.
Resultados
Kveik es un grupo genéticamente diferente de levaduras de cerveza
Para determinar si las muestras kveik originales contienen múltiples cepas de levadura, las muestras kveik se sembraron primero en agar WLN, que es un medio diferencial que permite distinguir Saccharomyces sobre la base de las diferencias en la morfología de la colonia y la absorción del colorante verde de bromocresol (Hutzler et al., 2015). Encontramos que todas menos dos de las muestras de kveik contenían más de una morfología de colonia de levadura distinta, que correspondía a cepas potencialmente únicas. El número de cepas aisladas de cultivos kveik individuales varió de 1 a 9 y ascendió a 25 y se resume en la Tabla 1.
Dado que los informes anecdóticos indican que las levaduras kveik son a menudo floculantes, demuestran una tasa de fermentación rápida y son capaces de utilizar azúcares de malta, todos los cuales son características de la domesticación (Gallone et al., 2016), nuestro objetivo fue determinar los parientes cercanos más cercanos levaduras kveik entre cepas conocidas de S. cerevisiae, y para determinar si las levaduras kveik están relacionadas entre sí. Como casi todas las levaduras ale domesticadas pertenecen a la especie S. cerevisiae, planteamos la hipótesis de que los aislamientos kveik también pertenecen a S. cerevisiae (Almeida et al., 2015; Gallone et al., 2016; Gonçalves et al., 2016). Realizamos su secuenciación y descubrimos que todas las cepas kveik excepto una se identificaron (mediante búsqueda BLAST) como S. cerevisiae (Tabla 1). Encontramos que la cepa que se origina en Muri es más estrechamente homóloga a los híbridos triples de S. cerevisiae / eubayanus / uvarum previamente identificados, presentando esta cepa de levadura en particular como un híbrido domesticado de potencial intrigante que justifica una investigación adicional (Tabla 1).
Dado que las levaduras kveik parecen ser cepas de S. cevevisiae, seguidamente preguntamos cómo se relacionan genéticamente con otras levaduras de S. cerevisiae. Para responder a esta pregunta, realizamos PCR interdelta usando los conjuntos de iniciadores δ12 / 21 y δ2 / 12 (Legras y Karst, 2003; Hutzler et al., 2015). Los elementos δ están separados por distancias amplificables en el genoma de S. cerevisiae y, en consecuencia, la PCR interdelta puede usarse para amplificar las regiones interdelta, que a su vez pueden usarse para identificar rápidamente levaduras con fines genéticos comparativos (Legras y Karst, 2003; Hutzler et al. al., 2015).
Ensayos preliminares usando los conjuntos de iniciadores δ1 / 2, δ2 / 12 y δ12 / 21 mostraron que los últimos dos conjuntos de cebadores producían el mayor rango de bandas útiles cuando se separaban mediante electroforesis en gel de agarosa. Luego ampliamos las regiones δ2 / 12 y δ12 / 21 de todas las cepas kveik y una selección de cepas de levadura que representan "Beer 1" (alemán, estadounidense, Reino Unido), "Beer 2" (Belgian Saison), sake, vino, pan , silvestres, y levaduras de destilación. La separación se realizó mediante electroforesis en gel capilar (Agilent Bioanalyzer), que proporcionó una mayor precisión y sensibilidad (Hutzler et al., 2015). El análisis de ambos conjuntos de datos δ2 / 12 y δ12 / 21 individualmente reveló que las levaduras kveik formaron un subgrupo entre las otras levaduras domesticadas, de modo que las levaduras kveik parecían estar más estrechamente relacionadas entre sí que con otras levaduras domesticadas (Figura S1 complementaria) . A continuación, creamos un análisis compuesto de los conjuntos de datos interdelta, produciendo un dendrograma que colocaba algunas cepas de cerveza muy juntas (Figura Suplementaria S2). Encontramos que un grupo de cepas de origen alemán, británico y estadounidense (WLP029, WLP002, WY1272, WLP007, BBY002) estaban representadas en el dendrograma y pueden representar el clado "Beer 1" (belga / alemán, británico, estadounidense), como fue identificado por Gallone et al. (2016). Sin embargo, las levaduras kveik formaron un grupo de levaduras relacionadas con un posible antecesor común. Las levaduras kveik parecen estar más relacionadas con las cepas de cerveza que otros grupos de levadura. Además, otras levaduras de este estudio, como la levadura híbrida Muri, una levadura de pan noruega (Idun) y la cepa de levadura lituana (Joniškelis) no parecen encajar dentro de la familia kveik. En conjunto, estos resultados sugieren que las levaduras kveik podrían representar un grupo genéticamente distinto de levaduras. Si bien no resuelve adecuadamente la filogenia debido a la falta de detalles, el método de huellas dactilares interdelta puede utilizarse para evaluar qué levaduras kveik están estrechamente relacionadas entre sí, y cuáles podrían seleccionarse para un posterior análisis de secuenciación de modo que se seleccione un rango representativo de cepas.
Para comprender mejor la genómica de kveik en relación con otras levaduras de S. cerevisiae, se secuenciaron genomas enteros de seis cepas kveik (Tabla 1) utilizando tecnología de 150 pares de pares de Illumina con una cobertura promedio de 472 × a 1,221 × (Tabla 2). Estas cepas se seleccionaron en base a los resultados de la toma de huellas dactilares de ADN para representar diferentes subgrupos de la familia kveik. Además, dos cepas de control (WLP001 y Vermont Ale) se secuenciaron e incluyeron en el análisis filogenético. Las distribuciones de citometría de flujo y de frecuencia alélica sugirieron que las seis cepas kveik eran tetraploides (Tabla 2, Figuras Suplementarias S3-S5). Sin embargo, 4/6 cepas mostraron aneuploidía debido a cromosomas CNV, y de particular interés, 3/6 cepas que contienen una copia adicional del cromosoma IX. Las cepas kveik también mostraron altos niveles de heterocigosidad, ya que el número de SNP heterocigóticos varió de ~ 54,000 a 68,000 (Tabla 2). La densidad de SNP heterocigotos fue relativamente uniforme en las cepas, con pocas regiones que hayan experimentado pérdida de heterocigosidad (Figura S6 complementaria).
A continuación, se intentó analizar los parámetros relevantes para la elaboración de cerveza de las levaduras kveik en la fermentación de cultivo puro. Como las culturas kveik noruegas parecen contener a menudo múltiples cepas de levadura, existe la posibilidad de que las cepas sean interdependientes. Por lo tanto, es importante determinar las características de fermentación de las cepas individuales, ya que las fermentaciones de cultivo individuales mostrarían si las cepas kveik individuales pueden fermentar adecuadamente la cerveza. La imposibilidad de hacerlo sugiere que existe una ventaja adaptada a la naturaleza de cepas múltiples de las culturas kveik. Además, nuestro objetivo fue confirmar informes anecdóticos de que estas levaduras presentan fases de latencia cortas y muestran una buena cinética de fermentación.
Realizamos fermentaciones de prueba usando las cepas kveik de cultivo puro, así como los controles de levadura ale industriales relevantes (WLP001, WLP002, WLP029, WLP570, White Labs). En particular, se eligió WLP001 porque es una de las variedades ale más populares para la producción de cerveza artesanal. Las fermentaciones se realizaron a 30 ° C, que se ha informado que es una temperatura típica para las cervezas fermentadas utilizando kveik (Garshol, 2015). Con el fin de evaluar la tasa de fermentación durante las primeras fases de la fermentación del mosto, controlamos la pérdida de CO2 en las fermentaciones a través del pesaje. Usando esta técnica, observamos que las curvas de fermentación para kveik eran a menudo favorables en comparación con la cepa de control con un tiempo de retardo de fermentación más corto observado en algunas de las cepas (Figuras 4A, B). De las cepas de control, WLP002 produjo la mayor cantidad de CO2 después de 24 h. Encontramos que 11 de las cepas kveik superaron a WLP002 a las 24 h, y la cepa de mejor rendimiento (Laerdal 2) produjo 70.6% más de CO2 en las primeras 24 h de fermentación (Figura 4B). Se realizó un ANOVA de una vía con la prueba post hoc de Tukey y se determinó que las cepas Laerdal 1 y Laerdal 2 eran significativamente más rápidas en este período a P <0,05 (hoja de datos complementarios 2). Después del período de fermentación y maduración de 12 días, también se midió la concentración de etanol de las cervezas usando HPLC. Las cepas de ale de control produjeron valores de etanol en el orden ascendente esperado: WLP002 (4.33 ± 0.64%), WLP029 (4.60 ± 0.72%), WLP001 (4.94 ± 0.25%), WLP570 (5.14 ± 0.29%). Encontramos que las levaduras kveik produjeron rendimientos de etanol esperados dentro del rango esperado para cepas de cerveza de S. cerevisiae, con rangos de atenuación aparentes que abarcan 60-90%, y rendimiento de etanol que varía de 4.01 ± 0.55 a 5.98 ± 0.32% (Figura 4C). No se observaron agrupaciones estadísticamente significativas entre los datos de etanol (hoja de datos complementarios 2). Los datos de control combinados con el rendimiento de etanol de las levaduras kveik en la fermentación de mosto indican que las levaduras kveik atenúan el mosto dentro del rango esperado de las cepas ale industriales domesticadas.
 |
| Tabla 3. Número estimado de copias de los genes vinculados al transporte de maltosa en las seis cepas kveik secuenciadas. |
 |
| Tabla 4. Metabolitos de sabor de fermentación (ppm) producidos por levaduras kveik durante la fermentación de mosto a 30 ° C medidos utilizando HS-SPME-GC-MS. |
 |
| Tabla 5. Polimorfismos de un solo nucleótido con pérdida de función en PAD1 y FDC1 en las seis cepas kveik secuenciadas. |
También analizamos la viabilidad de las esporas de las 6 levaduras kveik secuenciadas. Se observó viabilidad de esporas razonable (40,6-63,4%) en 5/6 de las cepas, con una cepa ("Stordal Ebbegarden 1") que muestra una baja viabilidad de las esporas (Tabla 2). Curiosamente, todas las cepas kveik secuenciadas contienen una mutación de pérdida de función en RMR1, una proteína requerida para la recombinación meiótica (Jordan et al., 2007). Esta mutación (726A> T que causa el codón de detención perdido) solo es homocigota en la cepa "Stordal Ebbegarden 1", lo que puede explicar por qué esta cepa demostró una baja viabilidad de las esporas.
Termotolerancia, tolerancia al etanol y floculación en Kveik
Dado que los ensayos de fermentación iniciales demostraron que las levaduras kveik son en gran parte POF y producen sabores de ésteres frutales deseables, seguidamente investigamos la tolerancia al estrés y la floculación de estas levaduras para determinar mejor su utilidad potencial y confirmar estas características adicionales de domesticación. Teniendo en cuenta los informes de fermentación a alta temperatura de cerveceros noruegos tradicionales (Nordland, 1969; Garshol, 2014), controlamos el crecimiento de las levaduras kveik junto con levaduras ale conocidas como cepas de control (WLP001; ale americana, WLP029; ale alemana, WLP570; Ale belga, WLP002; ale británica) bajo condiciones de crecimiento a temperatura normal y alta (30-45 ° C).
Encontramos que 19/25 cepas kveik crecieron a> 1.0 OD600 a 40 ° C, mientras que solo 1/4 de las cepas ale control (WLP570) crecieron a esta densidad óptica a 40 ° C (Tabla 6). Además, las cepas de 11/25 kveik crecieron a> 0.4 OD600 a 42 ° C, mientras que solo una de las cepas ale de control (WLP570) fue capaz de hacerlo. Sorprendentemente, 19/25 cepas kveik al menos duplicaron su densidad celular a 43 ° C con la densidad óptica máxima a esta temperatura observada para Laerdal 1 (OD 600 0.44). Curiosamente, una de las cepas de control (WLP570) también mostró crecimiento a 43 ° C (OD 600 0.39). Estos datos indican que la tolerancia a altas temperaturas es común entre las levaduras kveik, y que la tolerancia a altas temperaturas a menudo es limitada entre las cepas ale americanas / británicas / alemanas (Gallone et al., 2016). En particular, las cepas kveik mostraron un crecimiento de hasta 43 ° C, acercándose al límite teórico, y el umbral tecnológico superior actual para el crecimiento celular de S. cerevisiae (Caspeta et al., 2013, 2016; Caspeta y Nielsen, 2015). Todas las cepas no crecieron a 45 ° C (datos no mostrados). Varias mutaciones en la levadura se han relacionado con la termotolerancia mejorada. En general, las levaduras kveik cayeron en agrupaciones estadísticas entre las cepas WLP001 / WLP002 / WLP029 y WLP570 (hoja de datos complementarios 2). Hemos observado mutaciones heterocigóticas de pérdida de función en varios genes relevantes para la termotolerancia, incluyendo KEX1 (proteasa de muerte celular, cepas Kveik secuenciadas 4/6), LRG1 (proteína activadora de GTPasa específica de Rho1 y regulador negativo de la pared celular controlada por PKC vía de integridad; 6/6 cepas kveik secuenciadas), SWP82 (miembro del complejo de remodelación de la cromatina SWI / SNF; 1/6 cepas kveik secuenciadas), RPI1 (modula la integridad de la pared celular; 6/6 cepas kveik secuenciadas), IRA1 / IRA2 ( Proteínas activadoras de GTPasa y reguladores inhibidores de la vía RAS-cAMP; cepas kveik secuenciadas 6/6 y 1/6, respectivamente) y CDC25 (factor de intercambio nucleotídico de guanina unido a la membrana y activador de la vía RAS-cAMP; secuencia 4/6 kveik) cepas) (Jones et al., 1991; Lorberg et al., 2001; Puria et al., 2009; Wallace-Salinas et al., 2015; Satomura et al., 2016; Huang et al., 2018; Tabla complementaria S5 )
 |
| Tabla 6. Tolerancia a la termotolerancia y al etanol en levaduras kveik. |
La floculación es un sello distintivo de la domesticación de la levadura, ya que esta propiedad mejora la capacidad del cervecero para cosechar levadura mediante cultivo superior o inferior en el fermentador. Evaluamos la floculación de las levaduras kveik utilizando el método de absorbancia del método de análisis ASBC Yeast-11 Flocculence (ASBC, 2011). Las cepas de control produjeron valores de floculación esperados: por ejemplo, la cepa belga (WLP570) no es floculante (2%) y la cepa británica (WLP002) es altamente floculante (98%) (Figura 5). Observamos altos niveles de floculación entre las levaduras kveik, pero esta propiedad no fue universal: 12/24 cepas tuvieron valores de floculación> 80% (altamente floculante), mientras que otros mostraron floculación muy baja (<20%, 4 cepas). Curiosamente, en la mayoría de las muestras de kveik que contienen más de una cepa, al menos una de las cepas mostró altas tasas de floculación superiores al 80% (Figura 5). Es posible que en los cultivos kveik originales de S. cerevisiae, las levaduras sufran co-floculación y, en consecuencia, algunas cepas nunca desarrollaron o necesitaron esta función (Smukalla et al., 2008; Rossouw et al., 2015). No obstante, la alta incidencia de floculación eficiente entre las levaduras kveik es un apoyo adicional a estas levaduras han sido domesticadas. Las variaciones en el número de copias relacionadas con los genes de floculación (FLO) son comunes entre las levaduras domesticadas (Dunn et al., 2012; Bergström et al., 2014; Gallone et al., 2016; Steenwyk y Rokas, 2017). Tras el examen de los datos del WGS, observamos un alto grado de variación del número de copias en los genes FLO en las cepas kveik secuenciadas (Tabla 7). Notablemente, la única cepa con muy baja floculación analizada con secuenciación del genoma completo ("Hornindal 2", 12.3%) tuvo una eliminación completa de FLO1, conocido por ser un gen crítico que confiere el fenotipo floculento (Vidgren y Londesborough, 2011). La floculación de esta cepa fue significativamente menor (P <0.05) en comparación con la cepa Hornindal 1. También vale la pena señalar que todas las levaduras kveik secuenciadas llevan un 425A> G SNP en FLO8 que causa la pérdida de un codón de parada, restaurando la funcionalidad de FLO8, que está inactiva en la cepa de referencia S288c (Liu et al., 1996).
 |
| Tabla 7. Variación estimada del número de copias entre los genes de floculación (FLO) en kveik. |
Aquí presentamos evidencia que sugiere que levaduras kveik obtenidas de cerveceros noruegos representan un grupo previamente no descubierto de levaduras de cerveza genéticamente distintas y domesticadas, y que estas levaduras tienen atributos prometedores de producción de cerveza (Almeida et al., 2015; Baker et al., 2015; Gallone et al., 2016; Gonçalves et al., 2016). Nuestros datos de huellas dactilares PCR sugieren cepas de levadura kveik forman un grupo genéticamente distinto de levaduras ale. Además, el análisis de secuenciación completa del genoma de un grupo representativo de 6 cepas muestra que las levaduras kveik forman un grupo distinto probablemente relacionado con el clado "Beer 1" pero con posible ascendencia mixta cuando los haplotipos separados de las levaduras kveik se analizan por separado. La ascendencia mixta aparentemente conservada de kveik es interesante dado que las levaduras de cerveza de mosaico / origen mixto no son particularmente comunes entre ninguno de los principales grupos de levadura de cerveza (Gallone et al., 2016). Es importante destacar que nuestro análisis de las levaduras kveik noruegas sugiere que la presión de producción de alta frecuencia de la industrialización puede no ser necesaria para la domesticación de las levaduras de elaboración de cerveza.
Nuestra investigación de los atributos de producción de cerveza con ensayos de fermentación a pequeña escala, cribados fenotípicos y secuenciación del genoma reveló que la mayoría de las levaduras kveik noruegas metabolizan rápidamente los azúcares del mosto (con CNV relacionadas en genes relevantes para maltosa), son POF- (con pérdida de -funciones de función en PAD1 y FDC1), floculan de manera eficiente (con CNV en la FLO y genes relacionados), y son altamente tolerantes al etanol y termotolerantes (rasgos típicamente poligénicos). Los fenotipos de domesticación y los marcadores de domesticación genómica en kveik se alinean en gran medida con los de las levaduras de cerveza domesticadas previamente analizadas (Gallone et al., 2016; Gonçalves et al., 2016). Por lo tanto, parece que kveik se han domesticado de manera similar a las levaduras ale industrializadas modernas. El aumento de las tasas de producción de las primeras cervecerías industriales en los siglos XVII al XVIII se propuso anteriormente para proporcionar las bases para la domesticación de la levadura de cerveza (Gallone et al., 2016). Aquí mostramos que las levaduras kveik, sorprendentemente, tienen características de adaptación similares al entorno de fermentación de la cerveza a pesar de presumiblemente ser domesticadas por cerveceros de granja sin la presión de producción de alta frecuencia de un ambiente industrial de elaboración de cerveza (Gallone et al., 2016). Por lo tanto, es posible que la producción de cerveza de alta frecuencia asociada con la industrialización no sea el único mecanismo de adaptación que da como resultado las levaduras de cerveza domesticadas que se usan en la actualidad. Ya sea que se conozca o no la existencia de prácticas similares de elaboración de cerveza a pequeña escala, análogas al cultivo cervecero noruego de la granja, resultó en la domesticación de las cepas de levadura en la industrialización anterior a la cerveza 1. A medida que se dispone de más datos genómicos de levadura, es posible identificar levaduras que están más estrechamente relacionadas con kveik y comprender mejor la línea de tiempo de domesticación para estas levaduras y para otras levaduras de cerveza domesticadas.
Aproximadamente un tercio de las levaduras kveik no flocularon con alta eficiencia. Esto puede estar influenciado por el procedimiento utilizado por los cerveceros de la granja para cosechar levadura para volver a cosechar, incluida la recolección de al menos algunas de las células de levadura de fermentación superior donde la presión evolutiva para flocular sería menor. Por lo tanto, no es sorprendente que algunas cepas kveik flocuten de forma menos eficiente que otras. Sin embargo, kveik puede presentar un nuevo modelo para comprender la co-floculación de levadura dada la capacidad de alta floculación en algunos pero no todos los miembros de un cultivo mixto de levadura (p. Ej., La cultura Hornindal) (Nishihara et al., 2000; Stewart, 2015) .
Las fermentaciones de mosto revelaron que las cepas kveik producen una variedad de ésteres afrutados, con caproato de etilo, caprilato de etilo, decanoato de etilo y acetato de fenetilo por encima del umbral de detección (Tabla 3), lo que indica que estas levaduras pueden usarse para producir cervezas con carácter afrutado. Cómo se comparan las levaduras kveik con una gama más amplia de levaduras industriales de cerveza en términos de diversidad e intensidad de producción de sabor es actualmente desconocida y es una limitación del presente estudio. Hemos demostrado que las levaduras kveik ale tienen una amplia gama de valores de atenuación de mosto. Como estas levaduras son POF, un rasgo deseable para la mayoría de los estilos de cerveza (McMurrough et al., 1996), también podrían tener una amplia utilidad para la producción de cerveza, con la selección del cervecero de acuerdo con los valores objetivo deseados y los perfiles de sabor deseados .
Sorprendentemente, nuestro cribado fenotípico reveló la termotolerancia favorable y la tolerancia al etanol de estas levaduras en comparación con las levaduras de cerveza domesticadas conocidas. La adaptación al calor a largo plazo es particularmente relevante para los procesos de fermentación realizados a temperaturas elevadas, incluidos los utilizados para la producción industrial de bioetanol. Se han identificado múltiples procesos y objetivos moleculares y celulares en la adaptación de la levadura al calor. Un estudio previo que investigaba la adaptación de la levadura a ~ 40 ° C durante un período prolongado identificó SNP en genes relacionados con la reparación del ADN, la replicación, la composición de la membrana y la estructura de la membrana como marcadores genéticos específicos de la termotolerancia (Caspeta et al., 2013) . De forma similar, identificamos los SNP en: CDC25, IRA1 e IRA2, que son genes que regulan la ruta RAS / cAMP / PKA; RPI1 y LRG1, lo que afecta la integridad de la pared celular; y KEX1 y SWP82 (Puria et al., 2009; Wallace-Salinas et al., 2015; Peeters et al., 2017; Huang et al., 2018). Estas mutaciones podrían ayudar a la termotolerancia en kveik y ser rutas futuras para el desarrollo de levaduras termotolerantes. Esta característica también tiene una aplicación potencial en la elaboración, ya que la inoculación de mosto a temperaturas más altas (> 30 ° C) sin comprometer el sabor podría ayudar a limitar la costosa refrigeración necesaria para controlar las temperaturas de fermentación del mosto controladas a 18-22 ° C. (Hill, 2015).
También demostramos que la tolerancia al etanol, conocida por ser un rasgo poligénico y genéticamente complejo que involucra múltiples alelos, es una adaptación común de las levaduras kveik. Si bien las alteraciones genéticas únicas pueden aumentar de forma incremental la tolerancia al etanol, no se aproxima al fenotipo poligénico / multialélico (Lam et al., 2014; Snoek et al., 2016). Los ambientes con alto contenido de etanol generalmente alteran la estructura y la función de la membrana celular, y afectan el plegamiento de la proteína. No es sorprendente que los genes vinculados a la tolerancia al etanol a menudo se asocian con: estabilización de las paredes celulares y las membranas celulares; aumentar la capacidad de plegamiento de la proteína; mantener el gradiente electroquímico a través de la membrana plasmática; y mantener la función vacuolar por mencionar algunas (Lam et al., 2014; Snoek et al., 2016). Sorprendentemente, casi un tercio de las levaduras kveik informadas aquí podrían crecer en presencia de un 16% de etanol. En consecuencia, observamos mutaciones en genes vinculados a la tolerancia al etanol entre las cepas kveik secuenciadas, que comprenden AGP2, PCA1 y VPS70 (Teixeira et al., 2009; Voordeckers et al., 2015). Curiosamente, estas mutaciones fueron siempre heterocigotas. Dado el etanol y las tolerancias a altas temperaturas de las levaduras kveik, es posible que estas levaduras puedan beneficiar a las industrias de destilación y bioetanol cuando estos rasgos son deseados (Caspeta et al., 2016).
Ahora se sabe que todavía existe una selección más amplia de levaduras kveik noruegas tradicionales, y es posible que otras levaduras domesticadas o "nativas" puedan existir en otras ubicaciones geográficas más allá de Noruega. La secuenciación completa del genoma de levaduras kveik adicionales podría apoyar mejor los subgrupos geográficos sugeridos en el presente estudio. Además, un análisis detallado adicional de los cultivos kveik individuales (por ejemplo, el cribado de más colonias) puede revelar una mayor diversidad de cepas que la evidente aquí. La aparente naturaleza de mosaico de los genomas kveik también merece mayor investigación. Para dilucidar la ascendencia de las cepas kveik con más detalle, se podría aplicar el uso de secuenciación de lectura larga para mejorar la calidad y la longitud de los bloques de haplotipos durante la fase y ampliar el conjunto de datos del genoma, por ejemplo, con los 1.011 genomas de levadura publicados recientemente ( Peter et al., 2018), utilizado para el análisis filogenético y de la estructura de la población. Es posible que a través de una selección y secuencia fenotípica más detallada, particularmente usando tecnología de lectura prolongada, una gama más amplia de tales levaduras pueda dar como resultado una comprensión más amplia de la domesticación de levadura de cerveza dadas las notables diferencias entre producción de cerveza de granja (infrecuente, no comercial) vs. producción de cerveza industrial (frecuente, comercial).
Declaración de disponibilidad de datos
Los conjuntos de datos de la secuencia del genoma completo generados para este estudio se pueden encontrar en el número de BioProject del NCBI PRJNA473622 (https://www.ncbi.nlm.nih.gov/bioproject/PRJNA473622).
Contribuciones de autor
CT, RP y KK llevaron a cabo los experimentos descritos en este estudio. KK realizó un análisis bioinformático de los datos de la secuencia del genoma completo. RP, CT y GM diseñaron los experimentos. LG contribuyó con materiales introductorios y proporcionó cultivos de levadura. RP, KK y GM escribieron el manuscrito. Todos los autores leyeron y aprobaron el manuscrito final.
Esta investigación fue financiada por un NSERC Discovery (# 264792-400922) y otorgó OMAFRA-University of Guelph Gryphons LAAIR (LAAIR2017-5321).
Declaracion de conflicto de interes
Los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Expresiones de gratitud
Agradecemos a Cam Fryer y Royal City Brewing por la donación de cerveza utilizada en este estudio, y a Brian Gibson por la lectura crítica del manuscrito.
Material suplementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fmicb.2018.02137/full#supplementary-material
Referencias
- Almeida, P., Barbosa, R., Zalar, P., Imanishi, Y., Shimizu, K., Turchetti, B., et al. (2015). A population genomics insight into the Mediterranean origins of wine yeast domestication. Mol. Ecol. 24, 5412–5427. doi: 10.1111/mec.13341
- Andrews, S. (2010). FastQC: A Quality Control Tool for High Throughput Sequence Data. Babraham Institute.
- ASBC (2011). Yeast−11. ASBC Methods of Analysis, 1–2.
- Ausubel, F. M., Brent, R., Kingston, R. E., Moore, D. D., Seidman, J. G., Smith, J. A., et al. (2002). Short Protocols in Molecular Biology, 5th Edn. Hoboken, NJ: Wiley.
- Baker, E., Wang, B., Bellora, N., Peris, D., Hulfachor, A. B., Koshalek, J. A., et al. (2015). The genome sequence of Saccharomyces eubayanus and the domestication of lager-brewing yeasts. Mol. Biol. Evol. 32, 2818–2831. doi: 10.1093/molbev/msv168
- Benaglia, T., Chauveau, D., Hunter, D. R., and Young, D. (2009). mixtools : an R package for analyzing finite mixture models. J. Stat. Softw. 32, 1–29. doi: 10.18637/jss.v032.i06
- Bergström, A., Simpson, J. T., Salinas, F., Barré, B., Parts, L., Zia, A., et al. (2014). A high-definition view of functional genetic variation from natural yeast genomes. Mol. Biol. Evol. 31, 872–888. doi: 10.1093/molbev/msu037
- Boeva, V., Popova, T., Bleakley, K., Chiche, P., Cappo, J., Schleiermacher, G., et al. (2012). Control-FREEC: a tool for assessing copy number and allelic content using next-generation sequencing data. Bioinformatics 28, 423–425. doi: 10.1093/bioinformatics/btr670
- Bolger, A. M., Lohse, M., and Usadel, B. (2014). Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics 30, 2114–2120. doi: 10.1093/bioinformatics/btu170
- Brown, C. A., Murray, A. W., and Verstrepen, K. J. (2010). Rapid expansion and functional divergence of subtelomeric gene families in yeasts. Curr. Biol. 20, 895–903. doi: 10.1016/j.cub.2010.04.027
- Caspeta, L., Chen, Y., Ghiaci, P., Feizi, A., Buskov, S., Petranovic, D., et al. (2013). Altered sterol composition renders yeast thermotolerant. Science 944, 1–5. doi: 10.1126/science.1258137
- Caspeta, L., Chen, Y., and Nielsen, J. (2016). Thermotolerant yeasts selected by adaptive evolution express heat stress response at 30°C. Sci. Rep. 6:27003. doi: 10.1038/srep27003
- Caspeta, L., and Nielsen, J. (2015). Thermotolerant yeast strains adapted by laboratory evolution show trade-off at ancestral temperatures and preadaptation to other stresses. MBio 6:e00431–15. doi: 10.1128/mBio.00431-15
- Chiang, C., Layer, R. M., Faust, G. G., Lindberg, M. R., Rose, D. B., Garrison, E. P., et al. (2015). SpeedSeq: ultra-fast personal genome analysis and interpretation. Nat. Methods 12, 966–968. doi: 10.1038/nmeth.3505
- Cingolani, P., Platts, A., Wang, L. L., Coon, M., Nguyen, T., Wang, L., et al. (2012). A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3. Fly 6, 80–92. doi: 10.4161/fly.19695
- Comuzzo, P., Tat, L., Tonizzo, A., and Battistutta, F. (2006). Yeast derivatives (extracts and autolysates) in winemaking: release of volatile compounds and effects on wine aroma volatility. Food Chem. 99, 217–230. doi: 10.1016/j.foodchem.2005.06.049
- Dunn, B., Richter, C., Kvitek, D. J., Pugh, T., and Sherlock, G. (2012). Analysis of the Saccharomyces cerevisiae Pan genome reveals a pool of copy number variants distributed in diverse yeast strains from differing industrial enviroments. Genome Res. 22, 908–924. doi: 10.1101/gr.130310.111
- Earl, D. A., and vonHoldt, B. M. (2012). STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method. Conserv. Genet. Resour. 4, 359–361. doi: 10.1007/s12686-011-9548-7
- Engan, S. (1972). Organoleptic threshold values of some alcohols and esters in beer. J. Inst. Brew. 78, 33–36. doi: 10.1002/j.2050-0416.1972.tb03425.x
- Evanno, G., Regnaut, S., and Goudet, J. (2005). Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study. Mol. Ecol. 14, 2611–2620. doi: 10.1111/j.1365-294X.2005.02553.x
- Gallone, B., Steensels, J., Prahl, T., Soriaga, L., Saels, V., Herrera-Malaver, B., et al. (2016). Domestication and divergence of Saccharomyces cerevisiae beer yeasts. Cell 166, 1397.e16–1410.e16. doi: 10.1016/j.cell.2016.08.020
- García-Alcalde, F., Okonechnikov, K., Carbonell, J., Cruz, L. M., Götz, S., Tarazona, S., et al. (2012). Qualimap: evaluating next-generation sequencing alignment data. Bioinformatics 28, 2678–2679. doi: 10.1093/bioinformatics/bts503
- Garrison, E., and Marth, G. (2012). Haplotype-based variant detection from short-read sequencing. arXiv [Preprint]. arXiv:1207.3907, 9.
- Garshol, L. M. (2014). Brewing with Kveik. Available online at: http://www.garshol.priv.no/blog/291.html
- Garshol, L. M. (2015). Brewing Raw Ale in Hornidal. Available online at: http://www.garshol.priv.no/blog/342.html
- Gibson, B. R., Lawrence, S. J., Leclaire, J. P. R., Powell, C. D., and Smart, K. A. (2007). Yeast responses to stresses associated with industrial brewery handling. FEMS Microbiol. Rev. 31, 535–569. doi: 10.1111/j.1574-6976.2007.00076.x
- Gonçalves, M., Pontes, A., Almeida, P., Barbosa, R., Serra, M., Libkind, D., et al. (2016). Distinct domestication trajectories in top-fermenting beer yeasts and wine yeasts. Curr. Biol. 26, 2750–2761. doi: 10.1016/j.cub.2016.08.040
- Haase, S. B., and Reed, S. I. (2002). Improved flow cytometric analysis of the budding yeast cell cycle. Cell Cycle 1, 132–136. doi: 10.4161/cc.1.2.114
- Hahne, F., LeMeur, N., Brinkman, R. R., Ellis, B., Haaland, P., Sarkar, D., et al. (2009). flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics 10:106. doi: 10.1186/1471-2105-10-106
- Hasund, S. (1942). “Bygget som ‘ølkorn’ i det vestlandske havreområdet,” in Or Norges Bondesoge, Glytt og granskningar, Vol. 1 (Oslo), 208–226.
- Heras, J., Domínguez, C., Mata, E., Pascual, V., Lozano, C., Torres, C., et al. (2015). GelJ–a tool for analyzing DNA fingerprint gel images. BMC Bioinformatics 16:270. doi: 10.1186/s12859-015-0703-0
- Hill, A. E. (ed.). (2015). “Introduction to brewing microbiology,” in Brewing Microbiology (Cambridge: Woodhead Publishing), 27–29. doi: 10.1016/B978-1-78242-331-7.02001-3
- Huang, C., Lu, M., Chang, Y., and Li, W. (2018). Experimental evolution of yeast for high-temperature tolerance. Mol. Biol. Evol. 35, 1823–1839. doi: 10.1093/molbev/msy077
- Hutzler, M., Koob, J., Riedl, R., Schneiderbanger, H., Mueller-Auffermann, K., and Jacob, F. (2015). “Yeast identification and characterization,” in Brewing Microbiology, ed A. E. Hill (Cambridge: Woodhead Publishing), 65-104. doi: 10.1016/B978-1-78242-331-7.00005-8
- Iacobsen, C. (1935). Ølbrygning i Gamle Dage, Den gamle by's Aarbog. Aarhus: Købstadmuseet “Den Gamle By”.
- Jones, S., Vignais, M. L., and Broach, J. R. (1991). The CDC25 protein of Saccharomyces cerevisiae promotes exchange of guanine nucleotides bound to ras. Mol. Cell. Biol. 11, 2641–2646. doi: 10.1128/MCB.11.5.2641
- Jordan, P. W., Klein, F., and Leach, D. R. F. (2007). Novel roles for selected genes in meiotic DNA processing. PLoS Genet. 3, 2368–2380. doi: 10.1371/journal.pgen.0030222
- Kopecká, J., Němec, M., and Matoulková, D. (2016). Comparison of DNA-based techniques for differentiation of production strains of ale and lager brewing yeast. J. Appl. Microbiol. 120, 1561–1573. doi: 10.1111/jam.13116
- Kopelman, N. M., Mayzel, J., Jakobsson, M, Rosenberg, N. A., and Mayrose, I. (2015). Clumpak: a program for identifying clustering modes and packaging population structure inferences across K. Mol. Ecol. Resour. 15, 1179–1191. doi: 10.1111/1755-0998.12387
- Lam, F. H., Ghaderi, A., Fink, G. R., and Stephanopoulos, G. (2014). Engineering alcohol tolerance in yeast. Science 346, 71–75. doi: 10.1126/science.1257859
- Legras, J.-L., and Karst, F. (2003). Optimisation of interdelta analysis for Saccharomyces cerevisiae strain characterisation. FEMS Microbiol. Lett. 221, 249–255. doi: 10.1016/S0378-1097(03)00205-2
- Letunic, I., and Bork, P. (2016). Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic Acids Res. 44, W242–W245. doi: 10.1093/nar/gkw290
- Li, H. (2011). A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics 27, 2987–2993. doi: 10.1093/bioinformatics/btr509
- Li, H., Handsaker, B., Wysoker, A., Fennell, T., Ruan, J., Homer, N., et al. (2009). The sequence alignment/map format and SAMtools. Bioinformatics 25, 2078–2079. doi: 10.1093/bioinformatics/btp352
- Libkind, D., Hittinger, C. T., Valério, E., Gonçalves, C., Dover, J., Johnston, M., et al. (2011). Microbe domestication and the identification of the wild genetic stock of lager-brewing yeast. Proc. Natl. Acad. Sci. U. S. A. 108, 14539–14544. doi: 10.1073/pnas.1105430108.
- Liu, H., Styles, C. A., and Fink, G. R. (1996). Saccharomyces cerevisiae S288C has a mutation in FLO8, a gene required for filamentous growth. Genetics 144, 967–978.
- Lorberg, A., Schmitz, H. P., Jacoby, J., and Heinisch, J. (2001). Lrg1p functions as a putative GTPase-activating protein in the Pkc1p-mediated cell integrity pathway in Saccharomyces cerevisiae. Mol. Genet. Genomics 266, 514–526. doi: 10.1007/s004380100580
- Martin, M., Patterson, M., Garg, S., Fischer, S. O., Pisanti, N., Klau, G. W., et al. (2016). WhatsHap: fast and accurate read-based phasing. bioRxiv[Preprint]. 1–18. doi: 10.1101/085050
- McMurrough, I., Madigan, D., Donnelly, D., Hurley, J., Doyle, A., Hennigan, G., et al. (1996). Control of ferulic acid and 4-Vinyl guaiacol in brewing. J. Inst. Brewing 102, 327–332. doi: 10.1002/j.2050-0416.1996.tb00918.x
- Meilgaard, M. C. (1982). Prediction of flavor differences between beers from their chemical composition. J. Agric. Food Chem. 30, 1009–1017. doi: 10.1021/jf00114a002
- Minh, B. Q., Nguyen, M. A. T., and Von Haeseler, A. (2013). Ultrafast approximation for phylogenetic bootstrap. Mol. Biol. Evol. 30, 1188–1195. doi: 10.1093/molbev/mst024
- Mukai, N., Masaki, K., Fujii, T., and Iefuji, H. (2014). Single nucleotide polymorphisms of PAD1 and FDC1 show a positive relationship with ferulic acid decarboxylation ability among industrial yeasts used in alcoholic beverage production. J. Biosci. Bioeng. 118, 50–55. doi: 10.1016/j.jbiosc.2013.12.017
- Mukai, N., Masaki, K., Fujii, T., Kawamukai, M., and Iefuji, H. (2010). PAD1 and FDC1 are essential for the decarboxylation of phenylacrylic acids in Saccharomyces cerevisiae. J. Biosci. Bioeng. 109, 564–569. doi: 10.1016/j.jbiosc.2009.11.011
- Ness, F., Lavallee, F., Dubourdieu, D., Aigle, M., and Dulaub, L. (1993). Identification of yeast strains using the polymerase chain reaction. J. Sci. Food Agric. 6, 89–94. doi: 10.1002/jsfa.2740620113
- Nguyen, L. T., Schmidt, H. A., Von Haeseler, A., and Minh, B. Q. (2015). IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Mol. Biol. Evol. 32, 268–274. doi: 10.1093/molbev/msu300
- Nishihara, H., Kio, K., and Imamura, M. (2000). Possible mechanism of co- flocculation between non-flocculent yeasts. J. Inst. Brewing 106, 7–10. doi: 10.1002/j.2050-0416.2000.tb00034.x
- Nordland, O. (1969). Brewing and Beer Traditions in Norway. Oslo: The Norwegian Research Council for Science and the Humanities.
- Novo, M., Bigey, F., Beyne, E., Galeote, V., Gavory, F., Mallet, S., et al. (2009). Eukaryote-to-eukaryote gene transfer events revealed by the genome sequence of the wine yeast Saccharomyces cerevisiae EC1118. Proc. Natl. Acad. Sci. U.S.A. 106, 16333–16338. doi: 10.1073/pnas.0904673106
- Peeters, K., Van Leemputte, F., Fischer, B., Bonini, B. M., Quezada, H., Tsytlonok, M., et al. (2017). Fructose-1,6-bisphosphate couples glycolytic flux to activation of Ras. Nat. Commun. 8:922. doi: 10.1038/s41467-017-01019-z
- Peter, J., De Chiara, M., Friedrich, A., Yue, J.-X., Pflieger, D., Bergström, A., et al. (2018). Genome evolution across 1,011 Saccharomyces cerevisiae isolates. Nature 556, 339–344. doi: 10.1038/s41586-018-0030-5
- Pfeifer, B., Wittelsbürger, U., Ramos-Onsins, S. E., and Lercher, M. J. (2014). PopGenome: an efficient swiss army knife for population genomic analyses in R. Mol. Biol. Evol. 31, 1929–1936. doi: 10.1093/molbev/msu136
- Pham, T., Wimalasena, T., Box, W. G., Koivuranta, K., Storgårds, E., Smart, K., et al. (2011). Evaluation of ITS PCR and RFLP for differentiation and identification of brewing yeast and brewery “Wild” yeast contaminants. J. Inst. Brewing 117, 556–568. doi: 10.1002/j.2050-0416.2011.tb00504.x
- Pina-Martins, F., Silva, D. N., Fino, J., and Paulo, O. S. (2017). Structure_threader: an improved method for automation and parallelization of programs structure, fastStructure and MavericK on multicore CPU systems. Mol. Ecol. Resour. 17, e268–e274. doi: 10.1111/1755-0998.12702
- Pritchard, J. K., Stephens, M., and Donnelly, P. (2000). Inference of population structure using multilocus genotype data. Genetics 155, 945–959.
- Purcell, S., Neale, B., Todd-Brown, K., Thomas, L., Ferreira, M. A. R., Bender, D., et al. (2007). PLINK: a tool set for whole-genome association and population-based linkage analyses. Am. J. Hum. Genet. 81, 559–575. doi: 10.1086/519795
- Puria, R., Mannan, M. A. U., Chopra-Dewasthaly, R., and Ganesan, K. (2009). Critical role of RPI1 in the stress tolerance of yeast during ethanolic fermentation. FEMS Yeast Res. 9, 1161–1171. doi: 10.1111/j.1567-1364.2009.00549.x
- Quinlan, A. R., and Hall, I. M. (2010). BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics 26, 841–842. doi: 10.1093/bioinformatics/btq033
- Raj, A., Stephens, M., and Pritchard, J. K. (2014). fastSTRUCTURE: variational inference of population structure in large SNP data sets. Genetics 197, 573–589. doi: 10.1534/genetics.114.164350
- Räsänen, M. (1975). Vom Halm zum Fass. Helsinki: Kansatieteellinen arkisto.
- Rodriguez, M. E., Orozco, H., Cantoral, J. M., Matallana, E., and Aranda, A. (2014). Acetyltransferase SAS2 and sirtuin SIR2, respectively, control flocculation and biofilm formation in wine yeast. FEMS Yeast Res. 14, 845–857. doi: 10.1111/1567-1364.12173
- Rodriguez-Bencomo, J. J., Muñoz-González, C., Martín-Álvarez, P. J., Lázaro, E., Mancebo, R., Castañé, X., et al. (2012). Optimization of a HS-SPME-GC-MS procedure for beer volatile profiling using response surface methodology: application to follow aroma stability of beers under different storage conditions. Food Anal. Methods 5, 1386–1397. doi: 10.1007/s12161-012-9390-x
- Roe, C., Smith, D. E., Williamson, C. H. D., Aziz, M., Keim, P., Hepp, C. M., et al. (2016). NASP: an accurate, rapid method for the identification of SNPs in WGS datasets that supports flexible input and output formats. Microb. Genom. 2:e000074. doi: 10.1099/mgen.0.000074
- Rogers, C. M., Veatch, D., Covey, A., Staton, C., and Bochman, M. L. (2016). Terminal acidic shock inhibits sour beer bottle conditioning by Saccharomyces cerevisiae. Food Microbiol. 57, 151–158. doi: 10.1016/j.fm.2016.02.012
- Rossouw, D., Bagheri, B., Setati, M. E., and Bauer, F. F. (2015). Co-flocculation of yeast species, a new mechanism to govern population dynamics in microbial ecosystems. PLoS ONE 10:e0136249. doi: 10.1371/journal.pone.0136249
- Satomura, A., Miura, N., Kuroda, K., and Ueda, M. (2016). Reconstruction of thermotolerant yeast by one-point mutation identified through whole-genome analyses of adaptively-evolved strains. Sci. Rep. 6:23157. doi: 10.1038/srep23157
- Simpson, W. J., and Hammond, J. R. M. (1989). The response of brewing yeasts to acid washing. J. Inst. Brewing 95, 347–354. doi: 10.1002/j.2050-0416.1989.tb04642.x
- Smukalla, S., Caldara, M., Pochet, N., Beauvais, A., Guadagnini, S., Yan, C., et al. (2008). FLO1 is a variable green beard gene that drives biofilm-like cooperation in budding yeast. Cell 135, 726–737. doi: 10.1016/j.cell.2008.09.037
- Snoek, T., Verstrepen, K. J., and Voordeckers, K. (2016). How do yeast cells become tolerant to high ethanol concentrations? Curr. Genet. 62, 475–480. doi: 10.1007/s00294-015-0561-3
- Steensels, J., Snoek, T., Meersman, E., Nicolino, M. P., Voordeckers, K., and Verstrepen, K. J. (2014). Improving industrial yeast strains: exploiting natural and artificial diversity. FEMS Microbiol. Rev. 38, 947–995. doi: 10.1111/1574-6976.12073
- Steensels, J., and Verstrepen, K. J. (2014). Taming wild yeast: potential of conventional and nonconventional yeasts in industrial fermentations. Annu. Rev. Microbiol. 68, 61–80. doi: 10.1146/annurev-micro-091213-113025
- Steenwyk, J., and Rokas, A. (2017). Extensive copy number variation in fermentation-related genes among Saccharomyces cerevisiae wine strains. G3 7, 1475–1485. doi: 10.1534/g3.117.040105
- Stewart, G. G. (2015). Seduced by yeast. J. Am. Soc. Brewing Chem. 73, 1–21. doi: 10.1094/ASBCJ-2015-0202-01
- Strese, E. M., and Tollin, C. (2015). Humle - Det Gröna Guldet. Stockholm: Nordiska Museets Förlag.
- Teixeira, M. C., Raposo, L. R., Mira, N. P., Lourenco, A. B., and Sá-Correia, I. (2009). Genome-wide identification of Saccharomyces cerevisiae genes required for maximal tolerance to ethanol. Appl. Environ. Microbiol. 75, 5761–5772. doi: 10.1128/AEM.00845-09
- Verstrepen, K. J., Derdelinckx, G., Dufour, J. P., Winderickx, J., Thevelein, J. M., Pretorius, I. S., et al. (2003). Flavor-active esters: adding fruitiness to beer. J. Biosci. Bioeng. 96, 110–118. doi: 10.1016/S1389-1723(03)90112-5
- Vidgren, V., and Londesborough, J. (2011). 125th anniversary review: yeast flocculation and sedimentation in brewing. J. Inst. Brewing 117, 475–487. doi: 10.1002/j.2050-0416.2011.tb00495.x
- Visted, K., and Stigum, H. (1971). Vår Gamle Bondekultur, Bind 2, Oslo: Cappelen.
- Voordeckers, K., Kominek, J., Das, A., Espinosa-Cantú, A., De Maeyer, D., Arslan, A., et al. (2015). Adaptation to high ethanol reveals complex evolutionary pathways. PLoS Genet. 11:e1005635. doi: 10.1371/journal.pgen.1005635
- Wallace-Salinas, V., Brink, D. P., Ahrén, D., and Gorwa-Grauslund, M. F. (2015). Cell periphery-related proteins as major genomic targets behind the adaptive evolution of an industrial Saccharomyces cerevisiae strain to combined heat and hydrolysate stress. BMC Genomics 16:14. doi: 10.1186/s12864-015-1737-4
- Watanabe, D., Hashimoto, N., Mizuno, M., Zhou, Y., Akao, T., and Shimoi, H. (2013). Accelerated alcoholic fermentation caused by defective gene expression related to glucose derepression in Saccharomyces cerevisiae. Biosci. Biotechnol. Biochem. 77, 2255–2262. doi: 10.1271/bbb.130519
- Yue, J. X., Li, J., Aigrain, L., Hallin, J., Persson, K., Oliver, K., et al. (2017). Contrasting evolutionary genome dynamics between domesticated and wild yeasts. Nat. Genet. 49, 913–924. doi: 10.1038/ng.3847
- Zheng, X., Levine, D., Shen, J., Gogarten, S. M., Laurie, C., and Weir, B. S. (2012). A high-performance computing toolset for relatedness and principal component analysis of SNP data. Bioinformatics 28, 3326–3328. doi: 10.1093/bioinformatics/bts606
- Citation: Preiss R, Tyrawa C, Krogerus K, Garshol LM and van der Merwe G (2018) Traditional Norwegian Kveik Are a Genetically Distinct Group of Domesticated Saccharomyces cerevisiae Brewing Yeasts. Front. Microbiol. 9:2137. doi: 10.3389/fmicb.2018.02137
Edited by:
Isabel Sá-Correia, Universidade de Lisboa, Portugal
Reviewed by:
Jean-luc Legras, Institut National de la Recherche Agronomique (INRA), France
Jose Sampaio, Universidade Nova de Lisboa, Portugal
Eladio Barrio, Universitat de València, Spain
Copyright © 2018 Preiss, Tyrawa, Krogerus, Garshol and van der Merwe. This is an open-access article distributed under the terms of the Creative Commons Attribution License (CC BY). The use, distribution or reproduction in other forums is permitted, provided the original author(s) and the copyright owner(s) are credited and that the original publication in this journal is cited, in accordance with accepted academic practice. No use, distribution or reproduction is permitted which does not comply with these terms.
*Correspondence: George van der Merwe, gvanderm@uoguelph.ca